张弓课题组联合承启生物在Nucleic Acids Research发表最强翻译组学数据库
2017-11
文章来自:admin 阅读次数:4302
我院张弓教授课题组与深圳承启生物科技有限公司近日在核酸领域的顶级专业期刊Nucleic Acids Research(IF=10.16)联合发表了目前最强的翻译组学数据库,继续保持其在翻译组学方面的国际领先地位。
翻译组学是一门新兴学科,研究mRNA到蛋白质的翻译过程中的总体。该学科由我院张弓教授创立,其课题组开发了翻译组学中几乎所有的技术。其中,从总体上测定正在翻译的mRNA是翻译组学中最重要的研究内容之一,技术手段包括RNC-seq和Ribo-seq两种大规模测序方法,在生物学的方方面面几乎都有重要的应用,也是人类蛋白质组计划的核心支柱之一。但此类实验极其难做,世界上能开展此类研究的实验室极少,因此充分利用现有的翻译组测序数据进行挖掘就成为了世界生物学界十分现实的需求。然而现有数据集十分分散,查找和对比不易,而且各课题组的材料、测序方法、分析方法等都有很大差别,得出的数据无法直接比较。生物学科研人员一般不具有分析海量的测序原始数据的能力和条件,更加制约了对现有数据的利用。
张弓教授课题组针对以上问题,开发了TranslatomeDB(翻译组学数据库http://www.translatomedb.net/),收集了迄今为止13个物种的3857个翻译组学相关测序数据集,是目前最为全面的翻译组学数据库。各种检索和对比只需要动动鼠标即可轻松完成。
该数据库的强大之处远不止于此,还在于利用超高精度的FANSe3代算法对所有数据集进行统一的高精度、高容错的分析。尽管各数据集使用的仪器不同、测序设置不同,但都可以放到一起来比较。由张弓教授开发的FANSe系列算法是目前世界上精度最高、稳健性最强的大规模测序快速比对算法,其实验验证率接近100%,在多个临床紧急案例中发挥重要作用,张弓教授也因此被邀请做TED演讲。FANSe3是FANSe系列的第三代算法,是深圳承启生物科技有限公司的横向研发项目,其准确度不逊色于前代,而速度却提升30多倍。依托于承启生物所建立的商业化测序分析云平台Chi-Cloud(http://www.chi-biotech.com/index.php/fenxi),3857个数据集只用了一个多小时便全部分析完成,平均每个数据集分析时间仅1秒多。
更厉害的是,TranslatomeDB还允许用户上传自己的测序数据集(包括RNC-seq,Ribo-seq和相应的mRNA-seq),和数据库中已收录的大量数据进行统一分析,简单快捷、傻瓜式操作,无需占用任何本地计算资源,也不需要用户具备高深的生物信息学技能。这一切都是免费的,所有的计算资源均由深圳承启生物科技有限公司无偿捐赠。
数据库的建立、维护及与Chi-Cloud云平台对接的工作,由承启生物的专业IT团队完成。正是有了公司的强大IT团队的努力和强大的Chi-Cloud云平台支持,TranslatomeDB从开始研发到投稿仅用时不到半年,良好的校企联合是其快速突破专业顶级期刊的保障。TranslatomeDB数据库一经发表便得到了Nucleic Acids Research编辑的高度赞赏,审稿过程也极为顺利。一位审稿人对TranslatomeDB的审稿评价仅一句话:“ I believe this resource will be very useful for many scientists”(我相信这个资源将对许多科学家非常有用)!
我院助理研究员刘婉婷博士、本科生郑廷锴、承启生物程序员向伦平为共同第一作者,张弓教授为通讯作者。主要经费来自于承启生物的横向研发项目。
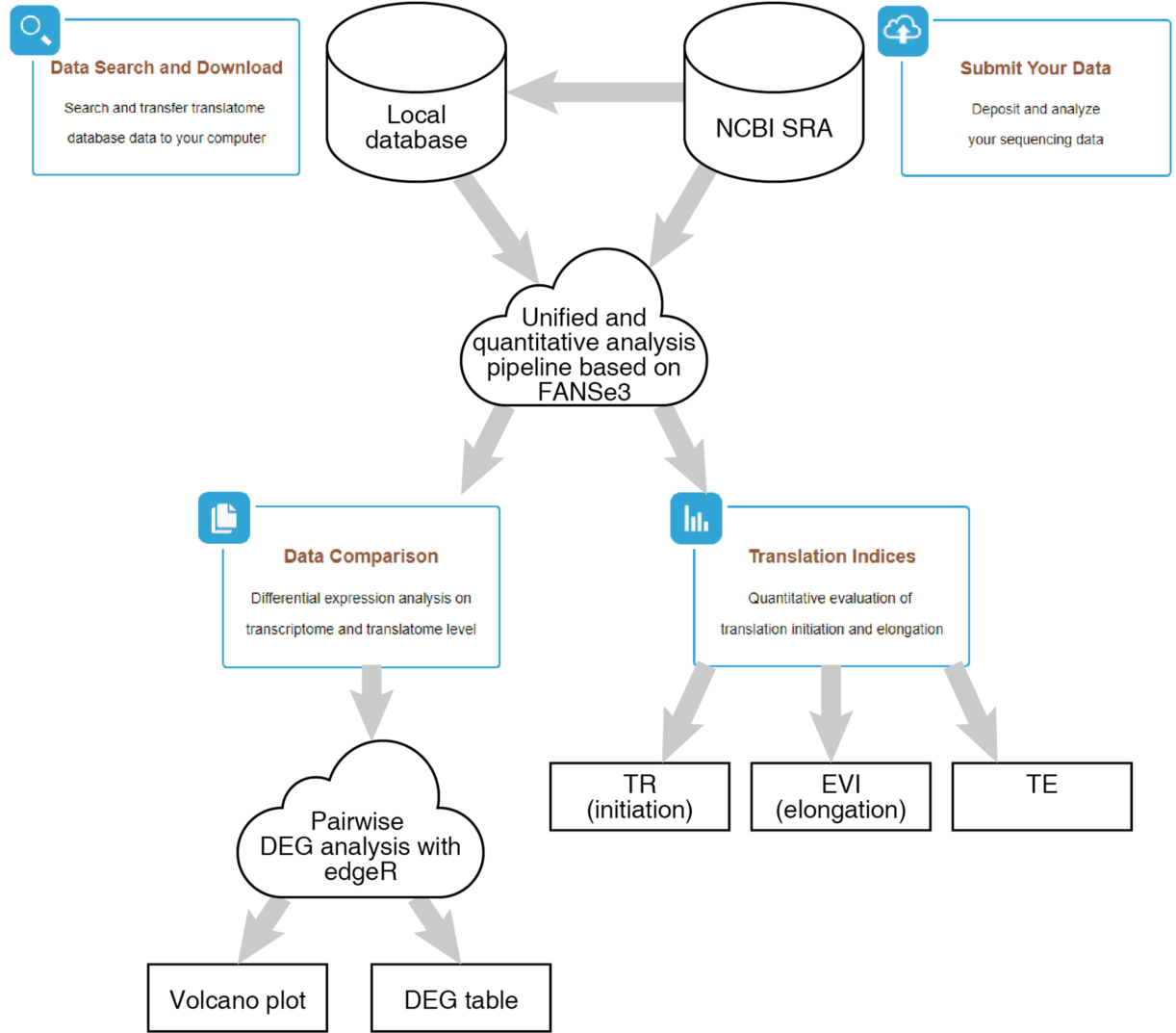
Functional flow chart of TranslatomeDB
文章链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkx1034/4584635

